平衡化
このステップで、統計力学的に意味のある構造に持っていきます。
前回
- 導入
- エネルギー最小化

1 相互作用パラメータ決め
最初のエネルギー最小化で作成した system.top をそのまま使います。
2 初期座標決め
エネルギー最小化処理で得た最後の構造 em.gro が初期構造となります。
3 MDの条件決め
以下のような内容で nvt.mdp というファイルを作成します。
integrator = md-vv
dt = 0.001
nsteps = 50000
nstenergy = 100
coulombtype = PME
gen_vel = yes
gen_temp = 300
tcoupl = nose-hoover
tc_grps = System
tau_t = 2.0
ref_t = 300
4 MD実行
まず読み込みを行います。
gmx grompp -c step1_em.gro -p system.top -f nvt.mdp -o step2_nvt.tpr端末の出力にNOTE (注意してほしいこと) が2つほどあるかもしれませんが、今回は無視します。これで step2_nvt.tpr というファイルができます。このままMDを実行します。
gmx mdrun -deffnm step2_nvt環境にもよりますが、数分待つかと思います。 これが終わると、以下のファイルが生成されています。
step2_nvt.cptstep2_nvt.edrstep2_nvt.grostep2_nvt.log
参考まで、以下の環境でテストを行いました。
- GPU:Tesla K80
- CPU:Xeon(R) CPU E5-2670 v3 @ 2.30GHz を 1コア利用
- GROMACS:2018
このとき、 step2_nvt.log の最後のほうに以下のような出力が得られました。
Core t (s) Wall t (s) (%)
Time: 49.165 49.165 100.0
(ns/day) (hour/ns)
Performance: 87.869 0.2735 結果の解釈
今回はNVTアンサンブルでの計算です。ということで温度がちゃんと設定した熱浴に収束しているか見てみようと思います。
gmx energy -f step2_nvt.edr -o temperature.xvg前回のエネルギー最小化のときと違い、 -o temperature.xvg というオプションが増えています。これは出力ファイル名をデフォルトの energy.xvg から temperature.xvg に変更するというものです。
これを実行すると、前回と同じように、どれを出力するかを指定するよう求められます。
Select the terms you want from the following list by
selecting either (part of) the name or the number or a combination.
End your selection with an empty line or a zero.
-------------------------------------------------------------------
1 LJ-(SR) 2 Coulomb-(SR) 3 Coul.-recip. 4 Potential
5 Kinetic-En. 6 Total-Energy 7 Conserved-En. 8 Temperature
9 Pressure 10 Vir-XX 11 Vir-XY 12 Vir-XZ
13 Vir-YX 14 Vir-YY 15 Vir-YZ 16 Vir-ZX
17 Vir-ZY 18 Vir-ZZ 19 Pres-XX 20 Pres-XY
21 Pres-XZ 22 Pres-YX 23 Pres-YY 24 Pres-YZ
25 Pres-ZX 26 Pres-ZY 27 Pres-ZZ 28 #Surf*SurfTen
29 T-System前回のエネルギー最小化のときと内容が異なり、4項目ほど増えていることに気づくかもしれません。このように、 gmx energy は読み込んだログファイルに応じて適切な項目を表示してくれます。
ここでは温度を見たいので Temperature に相当する 8 を選択します。
8 0と入力して Enter (または Return)キーを押します。するとこのコマンド実行により temperature.xvg が生成されていると思いますので、こちらを xmgrace などで可視化します。
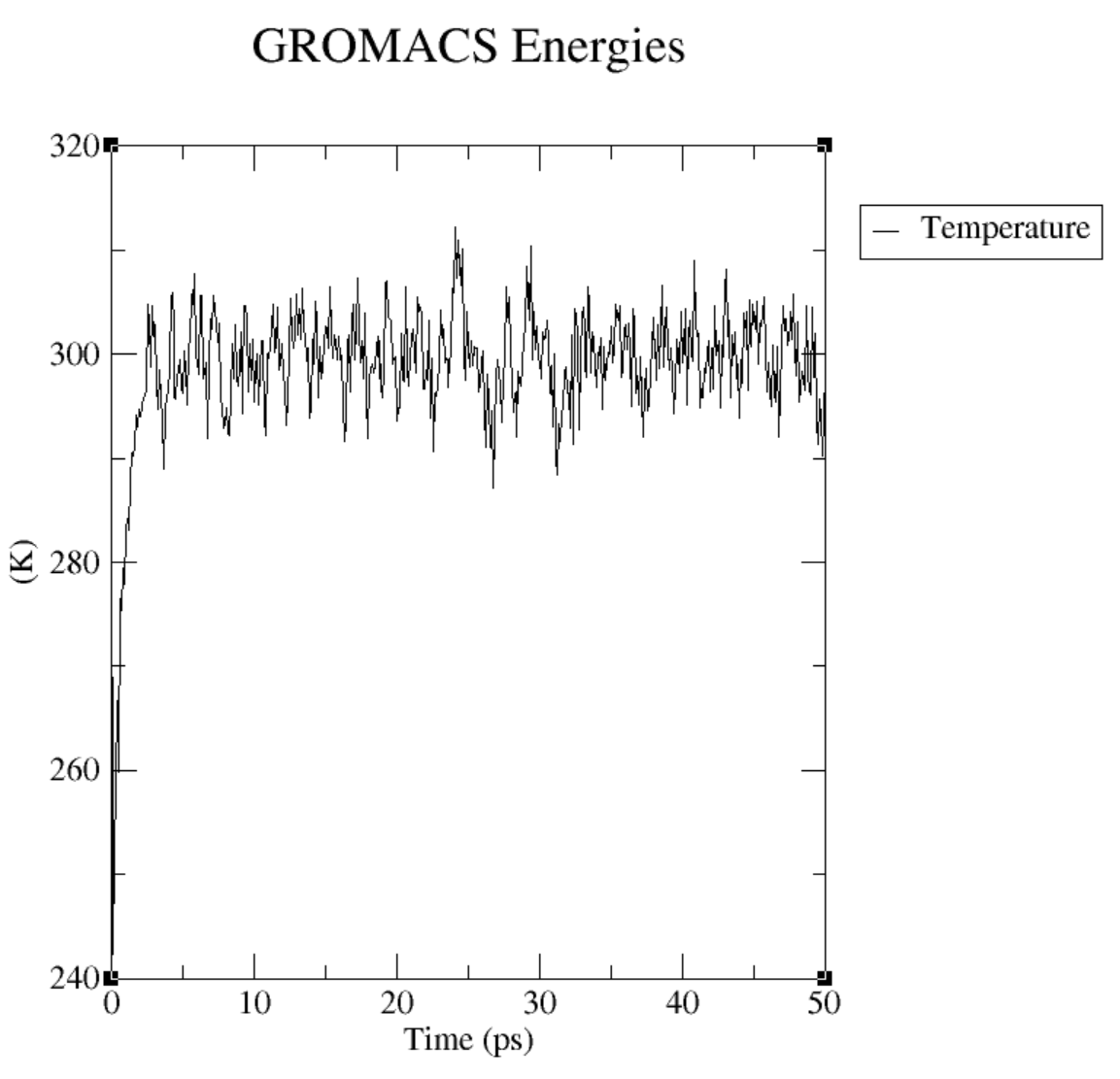 すぐに 300 K に落ち着き、その後は設定温度回りを振動しているのがわかります。
すぐに 300 K に落ち着き、その後は設定温度回りを振動しているのがわかります。
次のステップへ
厳密なことを言い始めると長くなるのですが、とりあえず今回のステップの最後で得られた構造 step2_nvt.gro を使ってサンプリングしてみます。

なお、次で最後です。
補足
エネルギー最小化で得られた構造ファイル step1_em.gro の中身はこんな感じです。
water in water
6495
1SOL OW 1 0.231 0.628 0.126
1SOL HW1 2 0.136 0.628 0.156
1SOL HW2 3 0.234 0.620 0.026
2SOL OW 4 0.225 0.284 0.987
2SOL HW1 5 0.255 0.253 1.077
3SOL HW2 6 0.130 0.255 0.971
以下略3行目以降に各原子の情報が並んでいます。前から順に
分子ID 原子種 原子ID x座標 y座標 z座標となっています。
一方でNVTアンサンブルの結果得られた構造ファイル step2_nvt.gro の中身は
water in water
6495
1SOL OW 1 0.217 0.536 3.984 -0.1820 -0.4114 -0.5572
1SOL HW1 2 0.228 0.635 3.992 -0.9125 -0.4490 0.9326
1SOL HW2 3 0.214 0.511 3.888 -1.0964 1.1462 -0.9353
2SOL OW 4 0.997 0.582 0.970 0.4162 -0.5560 0.6488
2SOL HW1 5 1.000 0.600 0.871 -0.0091 2.8599 1.2314
2SOL HW2 6 1.082 0.537 0.998 1.0203 -0.5205 -1.0919
以下略となっています。3行目以降は
分子ID 原子種 原子ID x座標 y座標 z座標 x速度 y速度 z速度という感じで、速度に関する情報が追加されているのがわかります。