導入事例 生命の基本原理を解明するために ~生命とは何か・なぜ多様な生物がいるのか~
東京大学
佐藤 直樹 様
"第18回目のインタビューは東京大学大学院総合文化研究科 広域化学専攻・生命環境化学系/広域システム化学系の教授を務めておられる佐藤直樹先生にお話をお伺いしました。
佐藤先生は、生物科学の課題である「生命とは無いか」と「なぜ多様な生物がいるのか」という2つの問を解明するために、計算(バイオインフォマティクス)と実験(分子生物学・生化学)の両面から研究を進められています。"
本日はよろしくお願いします。
– 早速ですが、はじめに佐藤研究室についてご紹介ください。
基本的には生物の研究室ですが、その中で生物情報科学(バイオインフォマティクス)を活用して新しい遺伝子機能を見つけたりするようなことを主にしています。
研究室全体は色々なことをやっていますが、特に光合成生物に関するゲノム学や代謝を調べています。最近では、藻類バイオに関係する研究も行っています。
佐藤先生とは、埼玉大学に勤務されていた2000年からお付き合いいただいていると伺っております。
確か、IA-64が出たばかりの頃でしたね。当時は、「それを活用して計算するのがこれからの主流だ」というイメージだったんですね(笑)。あっという間に変わってしまった訳ですけれども。はじめはそういうものからスタートしました。最初はDNAなどの配列を解析するためのごく簡単なツールを、自分で使い易いように作っていきました。
例えばDNAの配列からそれを翻訳したアミノ酸配列を作るとか。もちろんそういうものは既存のソフトにもありますが、その頃はありませんでした。それ以外にもDNAの特定の部分を切り出してくるとか。そういう時に、多くの生物学の人は画面上でコピーアンドペーストで作りますよね。それではやはりまずいだろうということで、すごく長い配列の中から何番目から何番目までの部分を持ってくるといったことが普通にできるようにしようということで始めました。
コンピュータを活用してDNAの配列を調べるといったことは、当時から盛んに行われていたのでしょうか?
もちろんある程度コンピュータは使われていました。SDC(Software Development Company)という会社が当時東京にありまして、そこで誰でも扱いやすいソフトが開発されていました。普通程度の遺伝子の配列をいじるのであればそういったソフトを使えば問題なく出来ました。ところが90年代途中ぐらいから「ゲノム配列」というものが世の中に出るようになってきまして、これは非常に大きな配列のため、簡単にいじれなくなってしまいました。「ゲノム配列を直接操作して、そこから必要な情報を得る」ということをするのが、最初の目的でした。
佐藤研究室では、主に光合成生物を題材にして、複数の研究テーマを持たれていますが、これらはすべて計算と実験の両面から研究されているということでしょうか?
すべてではありません。もちろん実験のほうがウエイトが大きいのですが、「新しい遺伝子を見つけ出す」という時に、極論を言うとそんなものは無い訳です(笑)。ヒトゲノムだってすでに遺伝子に番号がついています。しかし番号がついていて、こういう遺伝子の形のものということが分かっていても、それが「どういう働きをしているか」を調べるということが大事なのです。
例えば、シアノバクテリアという光合成をする微生物がいますが、そういったものを使って実験をする時に、ゲノムは読めていて、そこからどういう遺伝子があるかも大体推定出来ているとなると、配列としての遺伝子はあるけれどもそれが何をしているかという生物的な機能を明らかにするということはすぐには出来ません。「遺伝子の配列が似ている」ということを利用すれば、今ではかなりの遺伝子について「これが何のタンパク質に対応する」といったことが分かるようになりました。
しかし、私たちが研究し始めた2000年頃の時点ではまだ、遺伝子の配列がわかっていても、「実際にどういう酵素の遺伝子であるか」あるいは「何をしているタンパク質か」といったことはすぐには分からない状況でした。それからこの十数年間にずいぶんと発展があり、今ではどちらかというと分からないことのほうが少ないぐらいになりつつあります。ですが当時は分かるものが良いところ3割といった状況でしたので、「そういう機能を何割かでも見つかれば」ということでスタートした訳です。
光合成生物を対象にご研究に取り組まれるきっかけは何だったのでしょうか?
学生の時から光合成の研究室に属していまして、そもそも光合成というのは生物の世界で一番大切なことだと思うのです。もちろん普通の生物学で考えれば、人間のこととか、脳のことかが大事であるとは思いますが。
我々はご飯を食べて生きていますよね?お米は太陽の光を受けて育ちますから、結局生き物の世界は全部元を辿ると太陽の光のエネルギーで成り立っているということになる訳です。そう考えるとやはり「世の中に一番大事なのは光合成だ」と思いました。
子供の頃から科学にご興味があったのですか?
そうですね。子供の頃から実験をやっていました。小学生頃に「図書館の時間」というものがありまして、そこで実験の図鑑に巡り会いました。多分今の子供向けの本よりずっと危ない実験が書いてあったと思います。その中に青写真を作る実験が載っていまして。近くの薬屋さんで試薬を取り寄せて作ったりしていました。小学2年生ぐらいのことだったと思います。
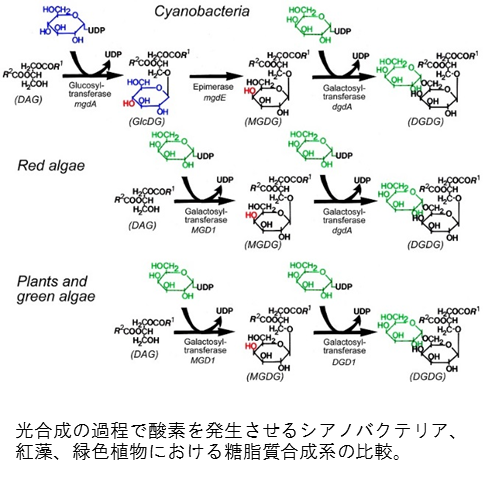
ご研究の話に戻りますが、UTokyo Researchに、先ほど触れられていたシアノバクテリアについて、その糖脂質合成経路の全容を解明されたことが掲載されていました。この糖脂質合成経路は約30年前に佐藤先生が博士論文で推定していたものだそうですね。
はい。そこに書いてある「代謝経路」というのは大学院の博士の研究の中で取り組んでいたものです。 代謝経路というのは放射性の同位元素を使って、それが最初の物質に入って、時間とともに他の物質に変換していくというような過程を見ているのですが、そういう実験をやりながら最初にこれができて、次にこれができてというような事を見て、そこからそういう経路を推定していました。でもそれはずっと昔の話なので、当時は遺伝子とかそういうことも何も分からない時代でした。シアノバクテリアがそういう代謝経路を持っているということだけが分かっていました。代謝経路には酵素があり、酵素をコードする遺伝子があるのですが、その遺伝子を実際に見つけない限り、それを証明したことにならない訳です。しかしそれはずいぶん後になってからです。私と一緒に研究した方に静岡大学の粟井さんという方がいらっしゃるのですが、その方がまだ静岡大学に来る前にシアノバクテリアの一個目の脂質合成の遺伝子を見つけてくれたんです。それはずっと昔に試験管内にこういう反応をやればできると分かっていたことでしたが、それが比較的やさしい訳なので、その遺伝子を彼が見つけて、3番目は私も見つけたのですが、その辺からコンピュータをうまく活用できるようになりました。
当時の計算環境ではこうした合成経路を網羅的に見るということはできなかったのでしょうか?
それは不可能ですね。当時御社から購入したコンピュータでも、確かメモリは16GBしか載りませんでした。今はタンパク質を比較する計算をしていますが、比較するタンパク質の数は200万個に及びます。これは16GBでは全然無理です。今は256GBのコンピュータがありますので、それを使えばかなり余裕で出来ます。
そんな無謀なことをそもそもやらなければいいという考えもありますけれども、結局比較というのはそれが一番簡単ですからね。そういうことを避けてコンピュータ的に良いやり方をうまく工夫するというのもありますが、それよりは直接的に比較するのが簡単ですのでそうしました。
ソフトウェアの開発はいつから取り組まれていることなのでしょうか?
90年代でしょうか、Macintoshのコンピュータは80年代ぐらいはまだ高嶺の花で、当時はNECのパソコンが一般的でした。博士論文の時の計算はNECのPC8000を使って行いました。その頃はN-BASICで計算をしていましたが、20×20の行列の計算をするだけで30分ぐらいかかったんですよ(笑)。そういうものを使って、C13という安定同位体でラベルする実験をやっていました。普通はC12ですから、C13が1個入ると質量が1だけ大きくなります。そういう調子でC13がたくさん入ってくると、どんどん重くなって分布ができるのです。
C13が入った分子がどの位あるかという分布を調べるために、質量分析から得られるデータをもとに行列式の計算をし、それでC13がいくつ入っているのかを正確に計算しました。それは今でも基本的には同じだと思いますが、そのようなことは私の中では、世界で最初に私がやったと思っていますが、質量分析の専門誌に当時、論文を出さなかったので、質量分析の業界では他の人がほとんど同じようなことをのちに論文に書かれています。10年ぐらいたってからのことでした。
当時から先端的な研究に取り組まれていたんですね?
そうだと思います。人々は認めてくれていませんけどね(笑)。質量分析の結果の解析に計算を利用するということは当時からやっていました。時間がかかって本当に大変でした。今同じ計算をやり直すと0.1秒かからないのですよ(笑)
コンピュータが高速化されたことで、ご研究の幅が広がった部分はありますか?
例えば今ご説明した質量分析の解析に関して言いますと、最近もう一度ソフトを作り直してやっていますが、GSL(GNU Scientific Library)の中に行列の計算はみんな入っているので、それを取り込んで計算をするようにしています。すると自由度が増してきて、シミュレーションなどもできるようになりました。「こういうパターンでラベルが入っている時は、最終的にこういう実験結果になる」ということがわかるので、シミュレーションを何通りか行い、一番近いものを見つけ出すということが可能になってきました。
また、遺伝子を実際に発見するに至った例を挙げますと、ここではシアノバクテリアに共通に存在している酵素であるはずだということで、シアノバクテリアに共通に存在するタンパク質の遺伝子を見つけてくる訳です。基本的には論文にはゲノムの配列が出ますと、どういうタンパク質がありそうだというデータまで入っていいますので、それを利用してゲノムの比較をやります。しかし「こういう遺伝子がありそうだ」とわかっても、別にそれは配列が分かるだけで機能は分からないですから、「この生物にはこういう遺伝子がある」「こういうタンパク質が入っている」というような単なる配列だけの集まりでしかありません。
そういう生き物を沢山集めてきて、そこに含まれているタンパク質を全部一緒にして、その中でどれとどれが似ているということを元にしてクラスターを作ります。そこで、シアノバクテリアのタンパク質だけでできたクラスターを探します。しかもできるだけ沢山のシアノバクテリアが入っているようなクラスターを探してくる訳です。そうするとその中に今回見つけたようなものもありました。今は機能が全く分からないということではなくて、ちょっとだけは機能が分かりそうなことが書いてあります。例えばここでは「酸化還元をする酵素」という一応の手がかりがありました。酸化還元の能力がある酵素は一応、配列情報の中でなんとなく分かるようになっています。
今求めているような酵素の活性というのは、おそらく部分的に酸化をして、また還元をするというようなことをやっているはずでした。しかし実際にどういう物質をどのように反応させるのかは分かりません。シアノバクテリアに共通に存在していて、しかも酸化還元能力があるような酵素を数えると、もう2つ3つになりました。その中の一つについては、代謝経路で1番目の酵素の遺伝子が、実はすぐそばにあったんです。
だからきっとこれに違いないと思いました。そこまでいくと人間の勘というのがあって、100パーセントコンピュータだけでやる訳ではなくて、やはり実際に実験していく以上、あんまり空振りしたくないですからきっとこれだろうとことをやって、それを調べていって当たる訳です。
勘が働く瞬間はどういう時に訪れるものなのでしょうか?
それは結局、結果次第ですね。いくつか候補が出てきた時に「きっとこれだろう」というのは色々と判断する基準がありまして。同じような酵素で、関連している遺伝子はどのようになっているといった情報は結構ある訳です。そういう知識を元にして考えることです。
研究成果は、どういったことに活用されるのでしょうか?
このシアノバクテリアの代謝系の話というのは、あまりに基礎的過ぎて、即利用できるというものではありません。ただ、「光合成生物がどういう進化をしてきたか」ということに関して大きなクエスチョンを投げかけるものだと思っています。
ここで分かったのはシアノバクテリアの話ですが、普通の植物でも同じようにガラクトースを含む脂質ができるのです。最終産物は一緒なのですが、合成経路は全然違います。しかも実は、植物の葉緑体というのは、元はシアノバクテリアが細胞に入ってきてできたと一般に考えられています。
そうなるとシアノバクテリアで、こういう仕組みで作っていたガラクト脂質が、シアノバクテリアの葉緑体になった時にどうして違う仕組みで作るようになったのかは大きな謎だと思います。物質としては同じものを作りますが、その作り方をどうして変える必要があるのか、作り方を変える以上は別の遺伝子をどこかからもらってこないといけない。これはかなり謎ですね。そういうことで、「生き物の進化に関する謎がちょっと深まったかな」と、そういう感じだと思います。
とても奥深いテーマですね。
不思議ですよね。光合成生物には非常に多くの種類がありまして、一番根本的な仕組みはほぼ同じなのですが、それぞれちょっとずつ違った仕組みでやっていますよね?というのは、地球上には色んな環境がありますから、それぞれの環境に応じてそこで一番効率良く光合成をするように……例えば海の海岸に近い場所は緑色の藻類がいて、ちょっと深くなると赤っぽいとかそういう住み分けがありますよね?しかし元を辿ると結局ひとつのものになります。そこがすごく不思議なところで、どうやって多様化してきたのか、そういうことをゲノムの比較の中から推定するんです。光合成生物の比較ゲノム解析はそういうことが目的でやっています。
同じ比較ゲノムでも医学の分野では大分違います。病気とか人間の発達に関係した異常とか、そういうことを研究するためにやっていますので。大分状況が違うかと思います。
これまで取り組まれたご研究の中で、最も印象深いものはどのような研究でしょうか?
やっぱりこれ(シアノバクテリアの糖脂質合成経路の研究)ですね(笑)。大学院生の時にやりましたが、その後色々事情があって、違うことをやっている研究室に行ったりして、そのままその研究を継続する訳ではなかったものですから。しかも遺伝子がわかるようになったのはいずれにしてもずっと後のことですので。さらに世の中の人は2000年ぐらいになってからこの研究をやりだしました。
他の研究者は遺伝子の候補を考えて、片っ端から調べていましたが,ひと通りやり尽くして「どれでもない」ということになって、「もう分からない」となりました。ところが比較ゲノムの知識を使うと一撃でできたというところが嬉しい訳です。他の人が一所懸命やったのに出来なかったというところが不思議なところですよね。
30年前に推定していたものと答えが一いたしたというのは嬉しいですよね。
基本的には正しかったようで、めでたいです(笑)。これが人間に関わることとか、もっと重要なことだったら良いのでしょうけど、ちょっと趣味的な感じにしか見えないかもしれません。
他の研究に取り組まれている間、この「シアノバクテリアの糖脂質合成経路」のことは頭の片隅にずっとあったものだったのでしょうか?
そうですね。代謝経路は実験で推定しますので、全然間違っているかもしれません。予想外のことが起きている可能性があって。昔の研究が全然間違っていたということになると,面白くないじゃないですか?博士論文で一所懸命やっていたものが違っていたりすると。そういう人も本当のところをいうと結構多くいるだろうと思います。
代謝経路は明確ですからね。AがBになってCになってという話ですから、そこが違っているというのは一目瞭然なので、いずれはそれをちゃんとやれるといいなとは思っていました。ただかなり長い期間違うことをやっていましたから、この問題に戻ってきたのは10年ぐらい前からです。
最近では実験系研究室でも、実験結果を裏付ける理論計算の重要性が謳われていますが、佐藤先生はご研究を始めた時から計算の必要性を感じていたのですか?
私自身は元々、ここの大学の理科一類に入っていまして、化学とかそういう方向でやろうと思っていましたが、そのうちに生物関係のことがこれから盛んになるということで、生物学の研究を始めました。でも昔は、生物学は好きではありませんでした(笑)。だから生物学というよりは生化学ですね、化学に近い範囲でやっていまして、その中で数学が使えれば良いなと思っていました。
量子化学も学ばれたと伺いました。
生物化学科は、当時まだ化学と生物の先生が寄り集まってできたばかりの学科でした。ですから、授業では化学の授業を半分取りました。その中で量子化学も勉強していました。
プログラムはすべて先生が作られているのですか?
基本は全部私が書いています。私自身はそんなにプログラミングの教育を受けておりませんので、ちゃんとしたものが作れているかどうかは分かりませんが、計算ができれば良いのだということで(笑)
若い頃から計算をされていたことは、ご研究を進める上で役に立ちましたか?
そうですね。今の学生さんなら結構気楽にやるとは思うんですけどね。当時N-BASICで計算するのはそれなりには流行っていてオタクみたいな人がいましたけど(笑)
その先どのぐらい繋がるかですね。大学の授業ではFortranを教わりましたが、基本は似ていますね。Fortranを1週間ぐらい教わったのを使って、あとはN-BASICで計算していました。Cに移ったのは90年代でした。MacintoshでCodeWarriorというソフトがありまして。それでプログラムが書いたりしていました。……でも恐ろしいですよ。クラッシュするとコンピュータごとクラッシュするので、一々再起動することになります(笑)。今の計算だったらクラッシュしてもプロセスが終わるだけになってしまいますね。
そうやっているうちにLinuxがだんだん出てきました。94年ぐらいですかね。本格的に使えるLinuxが出てきたのは。ヨドバシカメラでお正月に特別安く売るセールがありまして、それで小さなノートパソコンを15万円ぐらいで買いました。でもLinuxを入れようとしたら入らないんですよ。色んなドライバーが必要で。とはいえ、なかなかそういうものはないんですよ。使えるようになるまで大分苦労した記憶があります。それからはそれを使って計算をするようになりました。
翻訳も含め沢山の書籍を出されていますが、ご研究の傍ら執筆活動をされるのは大変ではないですか?
それほど大変ではありません。翻訳に関しては、面白そうな本があれば訳していくことは大した作業ではないです。実はフランス語を結構学んできておりまして、授業でもフランス語を教えていますけれども、フランス語で書かれた化学文献というのは中々日本語には訳されないので、そういったものを訳したいと思い、一所懸命やっています。
日本のバイオインフォマティクスや比較ゲノム解析といった研究は、世界的に見て進んでいる方なのでしょうか?
ずいぶんやられています。例えば医科学研究所というところが東大にはありますし、京都大学の化学研究所には、有名な金久 實 先生という方がいて、精力的に活動されていました。それらのウェブサイトにはゲノム情報のアノテーションや相互比較など色々な情報がまとまっていて、多くの人が活用されています。もちろんアメリカやヨーロッパにも大きな研究所があって盛んに研究が行われていますが、その中でも日本のレベルは高いほうだと思います。
実験と計算は、同時進行でされていますか?
並行していると思います。計算が裏にありながら実験をやっていくのが良いと考えています。でもオーソドックスな生物学ではまだウケないのです(笑)。生物学は“定性的”といって、「この遺伝子は何をしている」と一言で言える性質を見つけることが大事なんです。「この遺伝子を破壊することで生物がこんな風になってしまった」ということを元にして、「その遺伝子の機能がこうだ」ということを読み解くというやり方です。でも今はシステムバイオロジーのように計算をやる生物学も出てきました。計算で今実際に起きていることをシミュレーションし、それを確かめるというものです。
「ある遺伝子を壊したことでこうなる」というのもどちらかというと文系的な発想で、論理的に考えるだけですよね。実際に本当にシミュレーションするとこの遺伝子を破壊しても実はそうならないかもしれないということがありえます。今までは遺伝子を直接壊すということだけできたものを、壊した時に何が起きるかをちゃんとシミュレーションするとか、そういうことと組み合わせてやっていかないといけないと思っています。これからの生物学は化学や物理と同じように計算をして実験をして両方を突き合わせていくというのが良いのではないかと私は思います。
最後に、これから研究室所属される学生の方に向けてメッセージをお願いします。
佐藤研究室では計算と実験を融合して研究をしていきます。実際にそれをどういう場面で使うかというのは色々なことがあり得ます。例えば進化の研究とか代謝の研究とか、それぞれ違うかもしれません。頭の中ではシミュレーションなり計算をして、それで何か証明ができるようなことを考えだし、実験をし、証明をしていくということができれば良いのではないかと思います。
本日は大変有意義なお話をたくさんお聞かせ頂きありがとうございました
佐藤 直樹 先生のプロフィール

- 研究者紹介:
東京大学 大学院総合文化研究科 広域科学専攻・生命環境科学系 佐藤直樹研究室 - 研究テーマ:
・新型シーケンサによる全ゲノム解読とゲノム進化解析
・クラミドモナスやシアノバクテリアの運動によるパターン形成
・オルガネラの細胞内不均一性による環境耐性の調節
・光合成生物の栄養と光による細胞周期制御(コケ,紅藻)
・安定同位体と質量分析を用いた代謝フロー解析
・核様体タンパク質である亜硫酸還元酵素による環境浄化
お問い合わせ
平日9:30~17:30 (土曜日、日曜日、祝祭日、年末年始、夏期休暇は、休日とさせていただきます。)